如何预测基因功能
原创 小维 迈维代谢MetWare 2023-12-29 09:00 发表于湖北
实验原理
研究的目的基因是一种具有特定功能的结构基因,在对其分析时,主要是针对基因的功能预测与结构分析,包括基因的核苷酸序列、蛋白质序列及结构。
方法1:NCBI 数据库
1.基因核苷酸序列获取
我们一般研究的基因序列,都是经过高通量测序完然后GO注释完成的,针对某个物种某个基因序列,都会有相应的基因数据库(基因组或者转录组)能让我们去获取基因序列(如果是没有数据库的物种,基因序列可能就需要通过同源克隆的手段克隆出基因然后单克隆测序确定基因的序列),在这里,我们以拟南芥糖基转移酶(AT2G43820)为例,介绍如何对研究的基因进行大致的分析(需要注意的是,我们针对基因所进行的功能分析是基于编码蛋白质的CDS序列而言,不针对基因genome序列),我们在tair(TAIR - Home Page (arabidopsis.org))网站上获取到基因信息,下载好基因的CDS序列和蛋白质序列(fasta格式)
2.基因翻译
如果说在某个数据库里面找到基因的CDS序列,但是不知道蛋白序列(因为有的CDS序列是从转录组提取的),我们可以通过在线网站对CDS翻译成蛋白质序列,操作如下,复制CDS全长(从ATG至TAG/TAA/TGA)
搜索网址:https://web.expasy.org/translate/
点击“TRANSLATE”之后,等结果出来,可以看到第一个结果是 从“M”到“-”这是翻译全长的蛋白质序列,复制保存到新的txt文件里,保存格式为fasta。

3.用翻译的蛋白质进行blast
为了预测研究基因的功能,我们在ncbi大数据库里面进行blast来进行蛋白质功能预测(可能有的基因已经GO注释功能,但是还是blast,与已经发表的功能蛋白进行比对,保证结果更加准确)。
1)进入NCBI官网
https://www.ncbi.nlm.nih.gov/, 点击BLAST
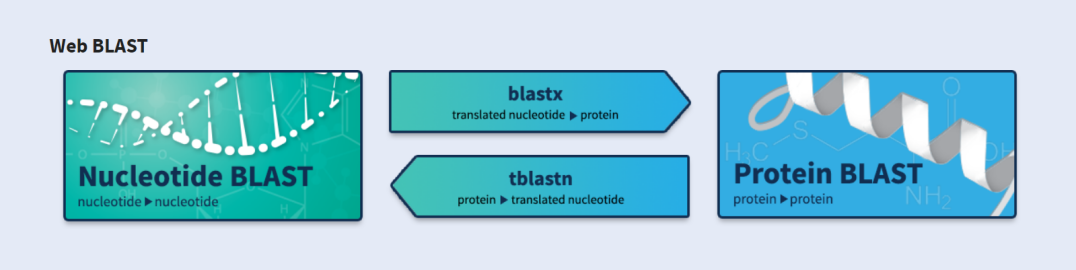
2)用候选基因蛋白序列进行比对,实操如下
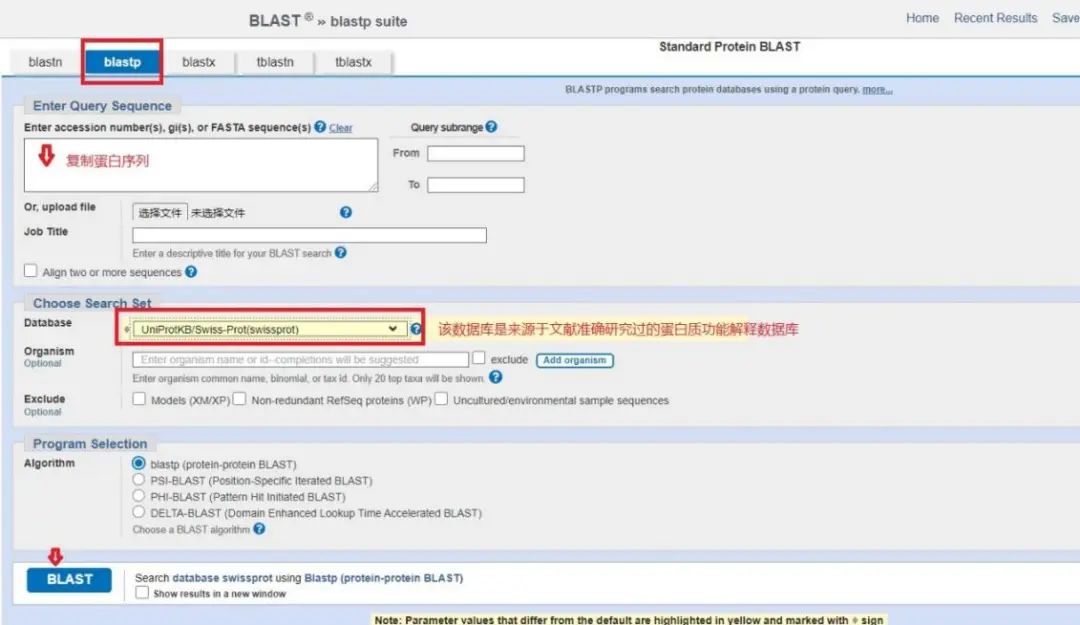
3)序列下载(还是以AT2G43820为例,这个基因我们从拟南芥数据库找到就是注释为糖基转移酶)
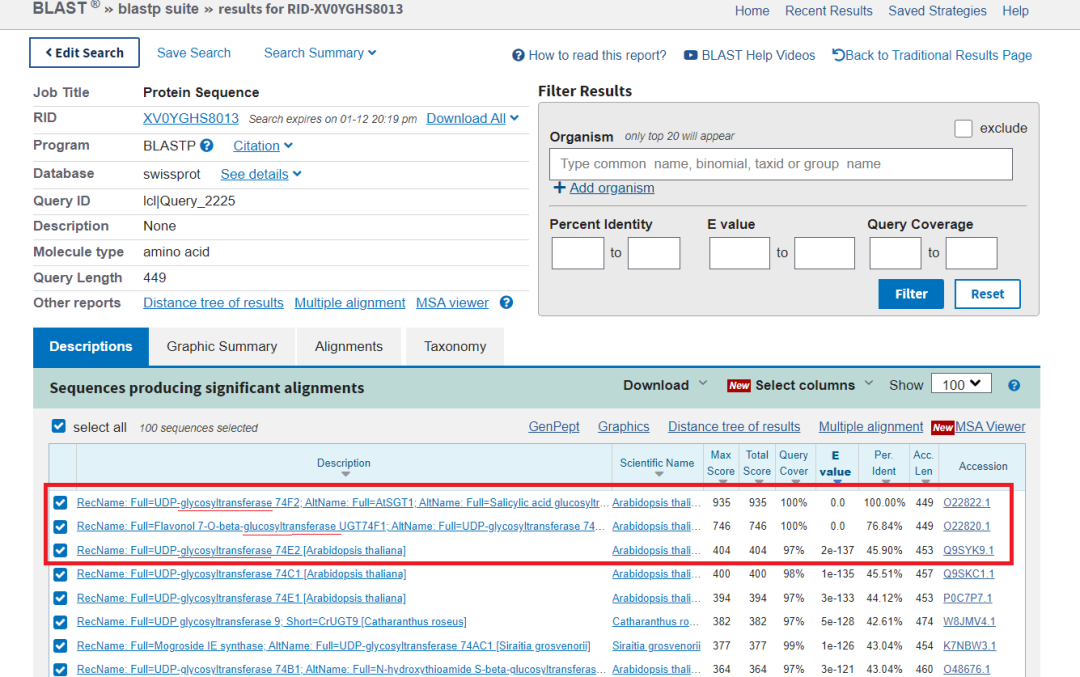
BlastP结果显示前面几条(就是E-Value较小的几个)都是报道过为GT(糖基转移酶)(到这里,如果结果都是表示为同一种功能蛋白,就已经说明研究的蛋白也是一种具有相同功能的蛋白),这里因为使用的是拟南芥基因,所以有2个blast结果E值为0,其实这就是AT2G43820的同源蛋白,我们按照未知的来处理,假设为研究蛋白blast出前3个结果,我们将这3个结果对应的蛋白序列都下载下来,保存为fasta格式。
Fasta格式类似于txt文本,只不过里面序列保存方式为:
>蛋白名称1(命名方式为英文字母或者数字)
>蛋白名称1(命名方式为英文字母或者数字)
>蛋白名称1(命名方式为英文字母或者数字)
…
选中需要下载的前几个序列,然后点击如图,批量下载文件:
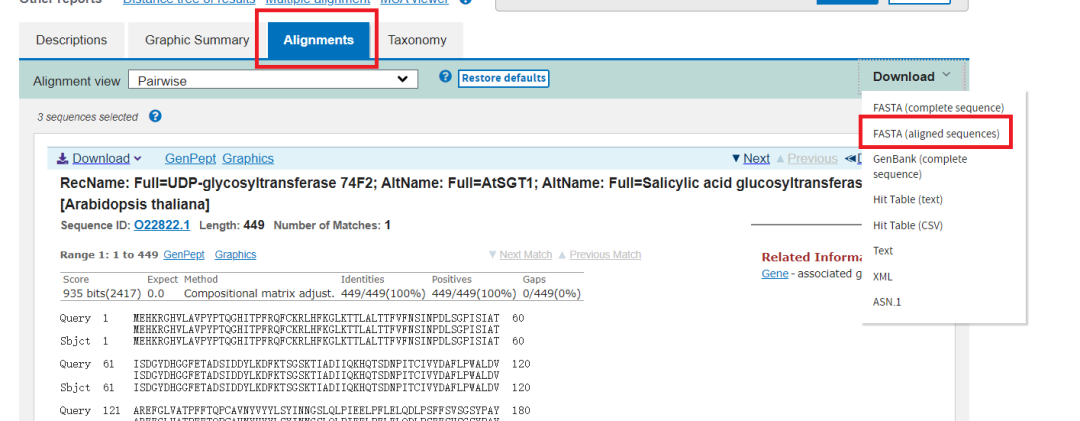
4.进行蛋白功能域分析
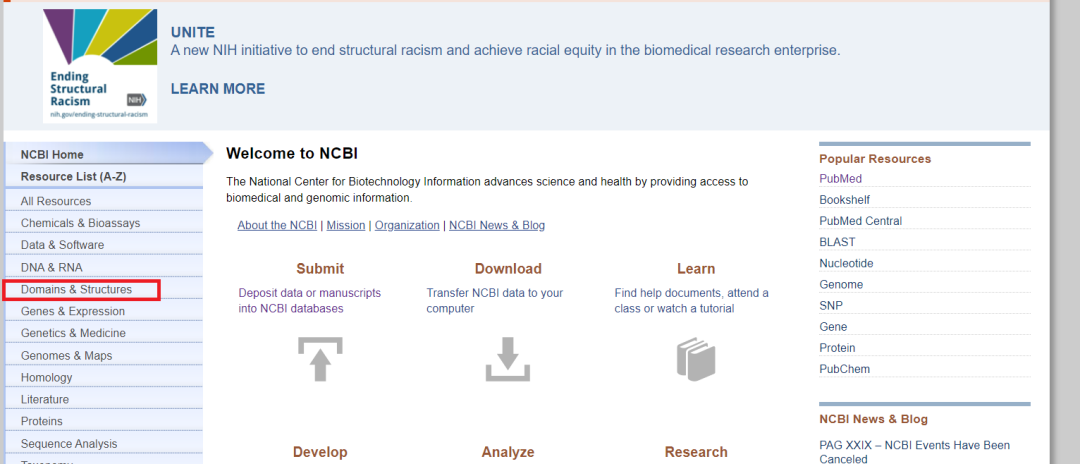
进入NCBI官网
https://www.ncbi.nlm.nih.gov/, 点击Domains &Structures。
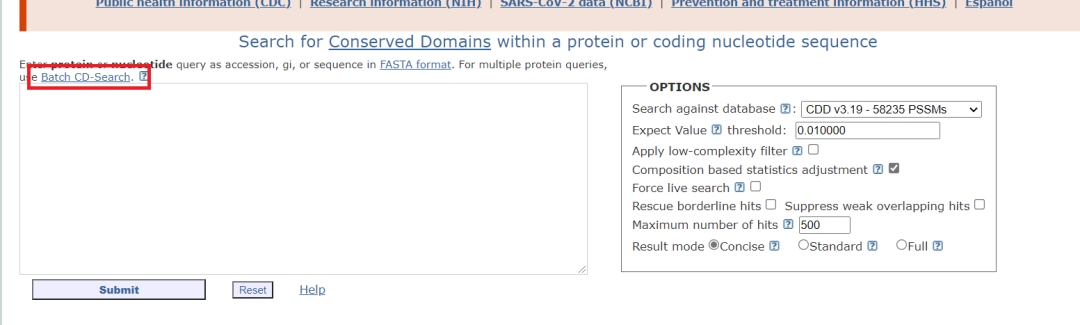
进入conserved Domain Database (CDD), 进入CD search,选择“Batch CD-search”,进入然后将下载好fasta的所有序列以及研究目的蛋白序列一起复制粘贴,然后点击“Submit”,开始对提交的蛋白质序列进行蛋白质保守功能域的查找。
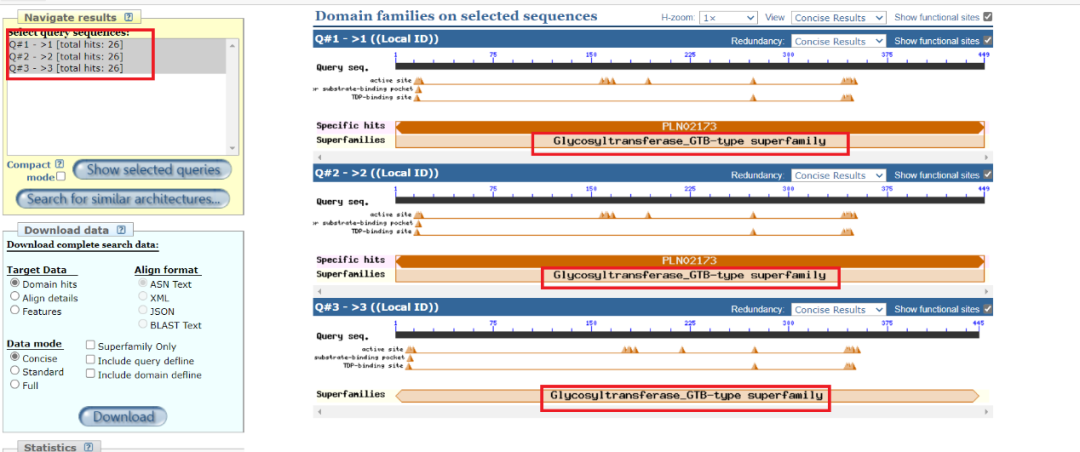
将搜索到的结构域结果全部展示,可以看到E值比较小的几个序列,所包含有的功能结果域基本相同,更加证明所研究基因“预测”所具有的功能是什么样的。
5.后续,根据预测的基因功能,查找相应的文献,总结体外酶活验证所需要的条件
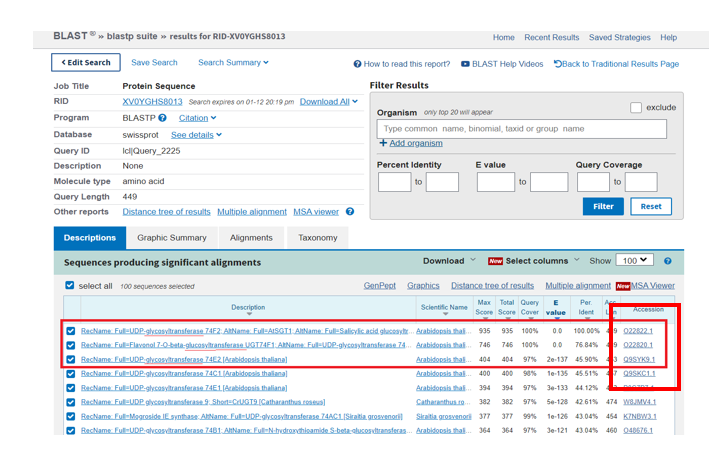
6.根据blast结果查阅相关文献,点击blast结果每行“Accession”
进入页面,就会有相关蛋白催化功能介绍,“COMMENT”栏会说明这个蛋白能够参与的反应途径,可能包括酶的功能介绍:从何种底物催化生成何种产物,或者参与某一类反应,或者会指出该蛋白属于哪类的酶家族,如EC=X.X.X.XX,此类
7.在页面每栏“Title”里面,会有相关文献记载
大致根据文献标题来判断是否设计该蛋白的酶活功能研究,根据标题搜索文献查看,具体为,在“Pubmed”数据库中输入文献名

点击进入,可查看做过的相关体外酶活实验。

方法2:Uniprot数据库使用
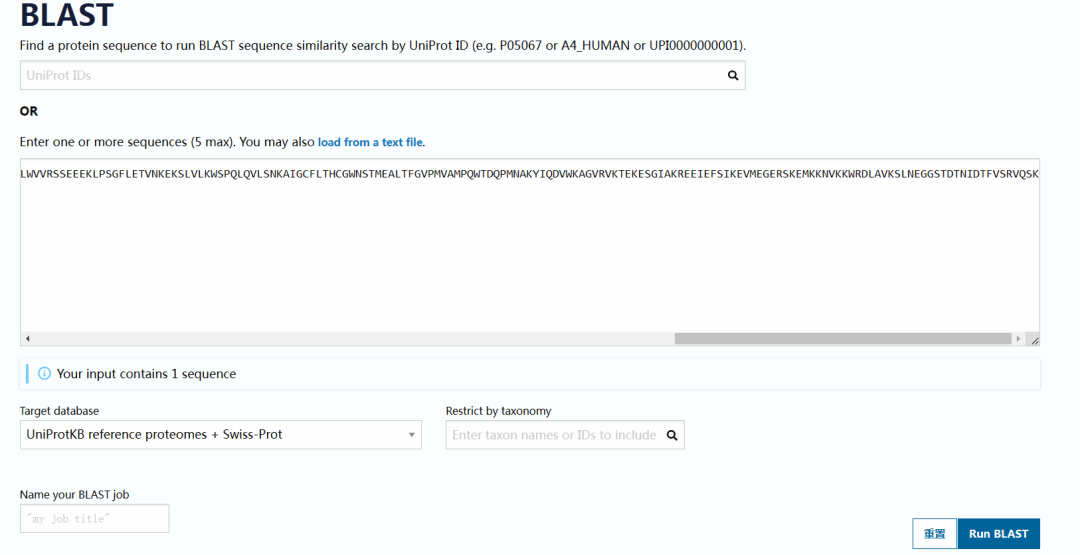
1.选择“Blast”程序,输入序列,点击“Run blast”
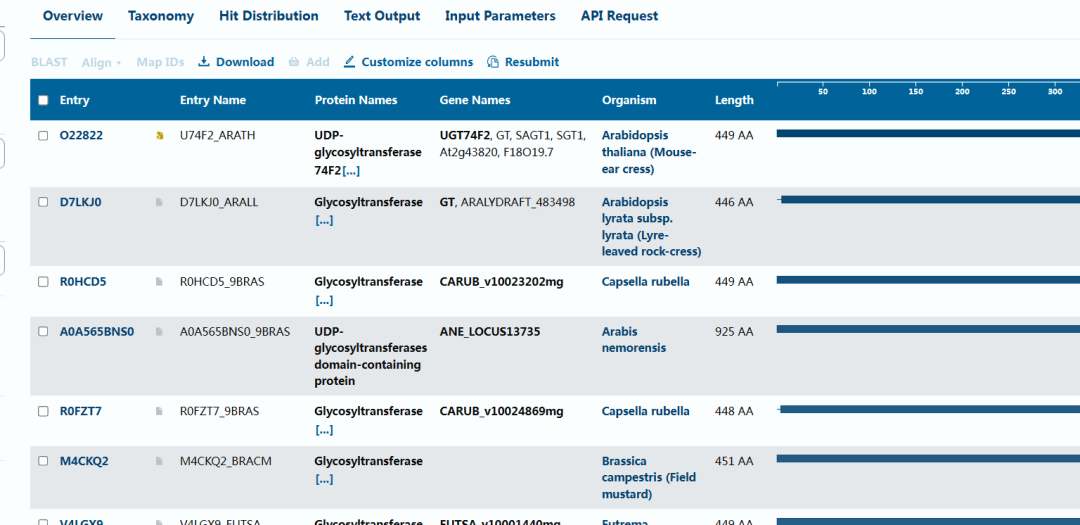
2.Blast结果,也是显示糖基转移酶,点击“Entry”列,每行进入查看
3.点击“publication”栏,查看相关功能研究文献
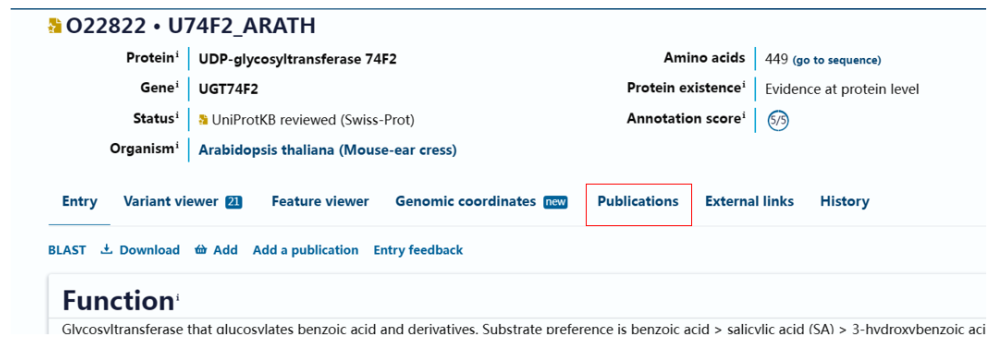
大致根据文献标题来判断是否设计该蛋白的酶活功能研究,根据标题搜索文献查看,具体为,在“Pubmed”数据库中输入文献名,点击进入,可查看做过的相关体外酶活实验。
Uniprot数据库使用,跟ncbi-blast中选择“UniprotKB/Swiss-Prot”相同,都是根据已报道过有功能记载的蛋白数据库进行匹配搜索。

